研究紹介
研究方針
近年の測定技術・データ処理技術の発達は、医療・保健分野にも多大な影響をもたらしています。例えば、ゲノムをはじめとするオミクスデータ、レセプトや電子カルテをはじめとするリアルワールドデータ等、大規模かつ多様な医療ビッグデータが蓄積されるようになりました。医療ビッグデータが増加する一方、そのデータを活用した新たな知見の発見、新たな治療法の開発が期待されています。
「実社会情報健康医療学講座」では、データサイエンス、生物統計、バイオインフォマティクス、疫学、公衆衛生学を駆使して医療ビッグデータを多角的に分析し、1.疾患発症や重症化に至る危険因子の探索、2.疾患発症・重症化予測アルゴリズムの開発を行い、これらを基に3.最適な予防策・介入策の提示を行い、医学・保健学分野の発展を目指しています。
また、研究活動を通してビッグデータ解析を取り扱える医療・保健学分野のデータサイエンティスト育成も進めています。
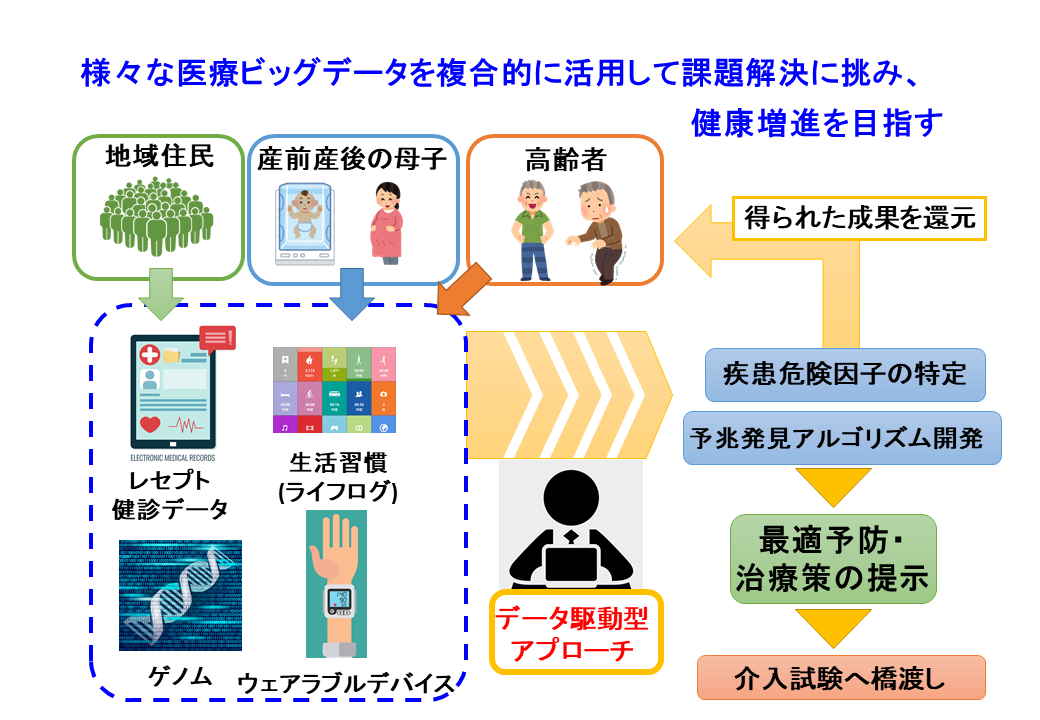
研究概要
「実社会情報健康医療学講座」では、ゲノム情報・レセプトや電子カルテをはじめとする大規模な医療・健康データ(ビッグデータ)を活用した研究を行い、疾患発症や重症化に至る危険因子の探索、疾患発症・重症化予測アルゴリズムの開発を進めています。
具体的な研究テーマとして、以下に示す医療・健康ビッグデータの解析研究を行います。
- 疾患や体質のゲノムデータ解析
- 血中のマルチオミクスデータを活用した疾患診断モデル・疾患発症予測モデルの開発
- 健診ビッグデータを用いた生活習慣病・精神疾患発症予兆予測システムの開発
- 母体・新生児データを用いた新生児の予後予測
- 健康診断データを用いた要介護認定に至るリスク要因の探索
- その他、現在公開されているor自分達が取得できるビッグデータで学生自身が行ってみたい医学・保健学的課題も推奨します。
1.疾患や体質のゲノムデータ解析
生まれつき背が高い人・低い人、お酒に強い人・弱い人、病気にかかり易い人・そうでない人というように、ヒトの体質には個人差があります。この個人差をもたらす原因として遺伝的要因が考えられています。当研究室では、日本国内、国外の研究機関と協力して、一塩基多型(SNP)やコピー数バリアント(CNV)をはじめとするゲノムデータを活用した遺伝要因の特定を進めています。これまでに、肥満、高血圧、糖尿病などの生活習慣病やすい臓がんをはじめとするがんの発症に寄与するSNP、精神疾患に寄与するCNVの同定を行ってきました。これらの遺伝情報を活用した、疾患発症リスク予測モデル、疾患進行予測モデルの開発も進めており、ゲノム情報に基づいたテーラーメイド医療の実現を目指しています。
<成果例>
Nakamura R, Tohnai G, Atsuta N, Matsuda Y et al. A genome-wide association study 1 identifies the GPM6A locus associated with age at onset in ALS. Communications Biology 8: 1720 (2025), DOI:10.1038/s42003-025-09168-4, リンク, プレスリリース
Nakatochi M, Kushima I et al. Copy number variations in RNF216 and postsynaptic membrane-associated genes are associated with bipolar disorder: a case-control study in the Japanese population. Psychiatry and Clinical Neurosciences 79(1): 12-20 (2025), DOI:10.1111/pcn.13752, リンク, プレスリリース
Koyanagi Y, Nakatochi M et al. Genetic architecture of alcohol consumption identified by a genotype-stratified GWAS, and impact on esophageal cancer risk in Japanese. Science Advances 10(4):eade2780 (2024), DOI:10.1126/sciadv.ade2780, リンク, プレスリリース, 名古屋大学フロントラインnote
Lin Y, Nakatochi M, Hosono Y, Ito H et al. Genome-wide association meta-analysis identifies GP2 gene risk variants for pancreatic cancer. Nature Communications 11: 3175 (2020), DOI:10.1038/s41467-020-16711-w, リンク
Nakatochi M et al. Genome-wide meta-analysis identifies multiple novel loci associated with serum uric acid levels in Japanese individuals. Communications Biology 2:115 (2019), DOI: 10.1038/s42003-019-0339-0, リンク
2.血中のマルチオミクスデータを活用した疾患診断モデル・疾患発症予測モデルの開発
近年の測定技術の発達により、様々な生体情報が網羅的に取得できるようになってきました。そのようなデータをオミクスデータと呼び、血液や様々な臓器からオミクスデータが取得され、研究されてきています。特に血液は体内をくまなく循環しており、また低侵襲性で繰り返し採取可能なことから、体内の異常を検知するためにオミクスデータの取得が盛んです。当研究室では、地域住民の血液から以下に示すような様々なオミクスデータを取得し、生活習慣病のようなありふれた疾患や筋萎縮性側索硬化症のような難病の早期診断や発症予測を可能とする因子の同定や、予測モデルの開発を進めています。
①DNAメチル化(エピゲノム)、②miRNA(トランスクリプトーム)、③タンパク質(プロテオーム)
<成果例>
Nakatochi M et al. Epigenome-wide association of myocardial infarction with DNA methylation sites at loci related to cardiovascular disease. Clinical Epigenetics 9:54 (2017), DOI: 10.1186/s13148-017-0353-3, リンク
Takemoto Y et al. Comparing preprocessing strategies for 3D-Gene microarray data of extracellular vesicle-derived miRNAs. BMC Bioinformatics 25: 211 (2024), DOI: 10.1186/s12859-024-05840-4, リンク
Beppo R and Ohashi Y et al. Evaluation of serum samples in long cryopreservation for SomaScan proteomics and sex differences in elderly Japanese adults. Scientific Reports 15: 29849 (2025), DOI: 10.1038/s41598-025-14464-4, リンク
Ohashi Y et al. Proteomic Footprint of Serum Urate Concentration and Urate Transporter ABCG2 Dysfunctional Polymorphism: A Cross-sectional Study. RMD Open 12: e006414 (2026), リンク
3.健診ビッグデータを用いた生活習慣病・精神疾患発症予兆予測システムの開発
生活習慣病・精神疾患発症は発症してから治療を開始するより、発症の予兆が出た段階から適切な介入を行うことが望ましいと考えられています。そこで、青森県弘前市岩城地区の住民の方々を対象に日々蓄積してきた健康診断データやゲノムデータを用いて、これらの疾患発症のリスク要因の探索や予兆を検知する予測モデルの開発を行っています。また健康寿命の延伸やWell-beingを実現するため、ビッグデータに基づくライフスタイルの策定も進めています。
プロジェクト ウェブサイト:https://coi.hirosaki-u.ac.jp/
<成果例>
Kinoshita F et al. Lifestyle parameters of Japanese agricultural and non-agricultural workers aged 60 years or older and less than 60 years: A cross-sectional observational study. PLoS ONE 18(10): e0290662 (2023), DOI: 10.1371/journal.pone.0290662, リンク
Oshima R and Ohashi Y et al. Longitudinal Trajectories of Health-Related Quality of Life and Their Predictors among Community-Dwelling Older Adults. Scientific Reports 16: 872 (2026), DOI: 10.1038/s41598-025-30307-8, リンク, プレスリリース
4.母体・新生児データを用いた新生児の産後予測
早産児の積極的な管理は、死亡率を改善させることが分かっています。そのためには、早産児の死亡リスクを出生前に正確に予測し、高リスク者を同定することが早期の新生児ケアのために必要不可欠です。3万人以上の日本人新生児とその母親のデータを基に、機械学習やAIを駆使して高リスクな早産児を同定するアルゴリズムの開発を行っています。
5.健康診断データを用いた要介護認定に至るリスク要因の探索
日本では2000年から介護保険制度が導入され、介護を必要とする人に介護サービスの費用を給付する制度ができました。日本では著しい人口高齢化が進んでおり、介護が必要であると認定された方は年々増加しています。要支援・要介護認定者の増加は財政圧迫や介護保険料の値上げ、介護施設への入所倍率の上昇など様々な問題を引き起こすため、介護予防介入による健康寿命の延伸が求められています。そのためには、要介護認定に至る前に適切な介入を行うことが重要であり、要介護認定の高リスク者を早期に特定する必要があります。当研究室では愛知県の複数の市町村から頂いた要介護認定情報や健康診断データをもとに、統計解析技術や機械学習・AIを駆使して要介護認定のリスク要因を探索し、高リスク者判定予測モデルの開発を行っています。
プロジェクト ウェブサイト:https://c-rex.thers.ac.jp/?page_id=35
<成果例>
Nakatochi M et al. U-shaped Link of Health Checkup Data and Need for Care using a Time-dependent Cox Regression Model with a Restricted Cubic Spline. Scientific Reports 13: 7537 (2023), DOI: 10.1038/s41598-023-33865-x, リンク, プレスリリース
Ohashi Y et al. Chronic Kidney Disease Progression, Long-term Nursing Care Burden, and Habitual Physical Activity: An Observational Study in Japan. BMJ Public Health 3(2): e003138 (2025), DOI: 10.1136/bmjph-2025-003138, リンク, プレスリリース
6.その他
現在公開されているor自分達が取得できるビッグデータで学生自身が行ってみたい医学・保健学的課題も推奨します。例えば、日本集中治療医学会が運営する日本ICU患者データベース(JIPAD)を用いてビッグデータ解析研究を大学院生が実施しました。JIPADのデータをもとに、手術後再挿管の発生率の推定やリスクマーカーの同定を行っています。今後も意欲のある保健学学生をヘルスデータサイエンティストとして育成していきます。
<成果例>
Masaki H et al. Risk markers for postoperative reintubation of intensive care unit patients: A retrospective multicentre study of the National Intensive Care Registry. Intensive and Critical Care Nursing 87: 103956 (2025), DOI:10.1126/sciadv.ade2780, リンク, プレスリリース, 名古屋大学フロントラインnote
データ解析支援(学術変革領域研究)
科研費受給者を対象に、研究を支援する体制を構築しています。その枠組みの中で当研究室が大規模データ解析の支援を実施しています。また、他のグループによる検体提供支援、オミクスデータ提供支援も行っています。皆さんの研究をより優れたものにするために、支援の活用をご検討ください。
プロジェクト ウェブサイト:https://square.umin.ac.jp/cohort/about/D.html